【Python_3D点群処理】3D点群データのクラスタリング (k-means, DBSCAN, HDBSCAN)
はじめに
今回は3D点群データのクラスタリングをします。クラスタリングとはデータ間の類似度に基づいてデータをグループ分けする手法です。
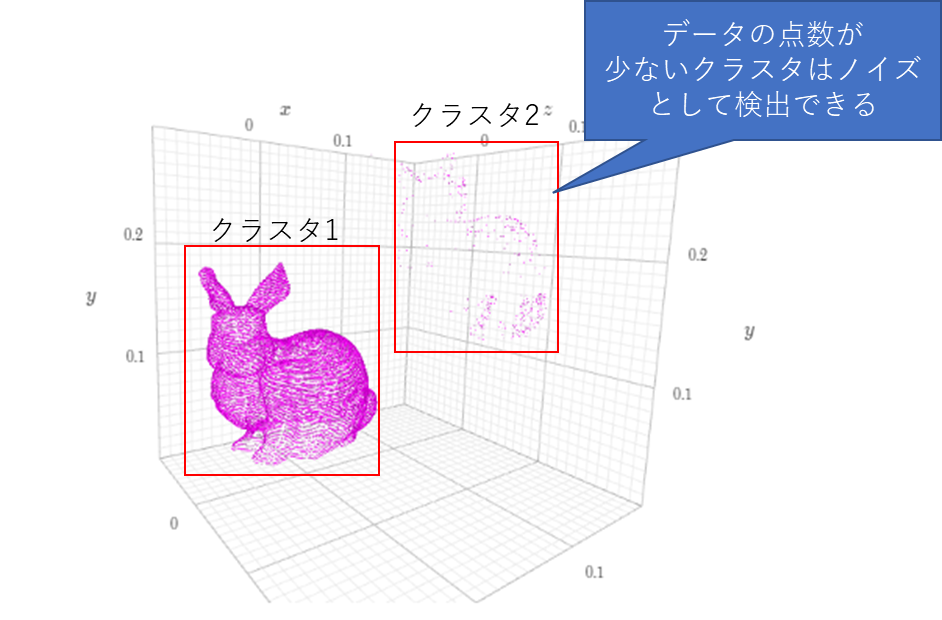
3D点群でのクラスタリングの応用先としては、①点群データ内の物体(クラスタ)の抽出や②ノイズ除去があります。
①クラスタリングで点群データのクラスタを抽出することで、クラス分類など後段の処理につなげることができます。

②クラスタリングを実施し、点の数が少ないクラスタはノイズとして削除することができます。
クラスタリングのアルゴリズムはいくつかありますが、今回はk-meansとDBSCAN、その発展形であるHDBSCANについての解説とPythonでの実装をします。
k-means
k-meansは、データを適当なクラスタに分けた後、クラスタの平均を用いてデータを分類していくアルゴリズムです。
設定するパラメータはクラスタ数kです。
アルゴリズム
1.あらかじめクラスタの数kを決める
2.ランダムな位置にクラスタの重心をK個定める
3.それぞれのクラスタの重心と各点の距離を計算
4.各点をもっとも近い重心に割り当て、同じ重心に割り当てられた点は同じクラスタとする
5.設定した回数もしくはクラスタの変更がなくなるまで3と4を繰り返す
Pythonコード
ライブラリのインポートをします。k-meansはsklearnに実装されています。
import numpy as np
import k3d
from sklearn.datasets import make_blobs
from sklearn.cluster import KMeans
import timek3dでプロットをするときに色を16進数で指定するので、ランダムに色を作成する関数colorsを定義します。
def colors(k):
ns = []
while len(ns) < k:
n = "0x" + "".join([random.choice("01234567789ABCDEF") for j in range(6)])
if not n in ns:
ns.append(n)
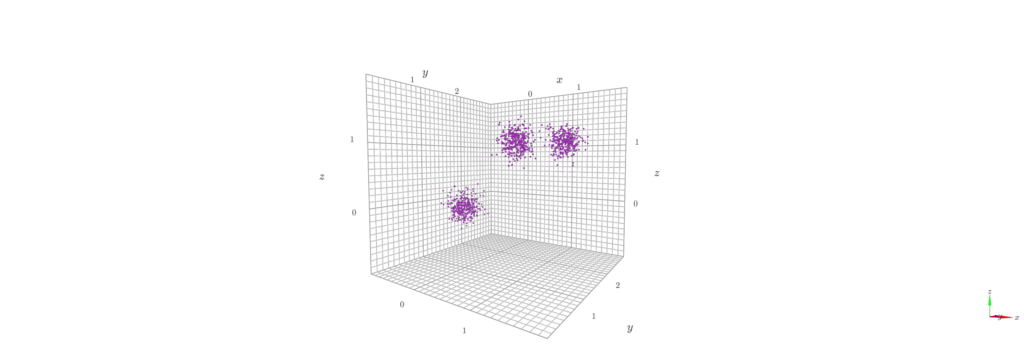
return nsクラスタリング用のデータを作成します。今回は1000個のサンプルを作成しました。
centers = [[0, 1, 0], [1.5, 1.5, 1], [1, 1, 1]]
stds = [0.13, 0.12, 0.12]
X, labels_true = make_blobs(n_samples=1000, centers=centers, cluster_std=stds, random_state=0)
point_indices = np.arange(1000)作成した点群データXです。
次に作成したデータXに対してk-meansでクラスタリングを実施します。今回はクラスタ数:n_clustersを3と設定しました。
n_clusters = 3
start = time.time()
kmeans = KMeans(n_clusters=n_clusters, random_state=9).fit(X)
result = kmeans.labels_
elapsed_time = time.time() - start
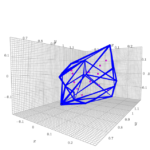
print ("elapsed_time:{0}".format(elapsed_time) + "[sec]")最後にクラスタリング結果を可視化します。クラスタごとに色を分けてプロットしていきます。
col = colors(100)
plot = k3d.plot()
for i in range(n_clusters):
plot +=k3d.points(X[result==i], point_size=0.04, color = int(col[i],16))
plot.display()うまく3つのクラスタに分類することができました。

DBSCAN
DBSCANは全てのデータを「ノイズ」か「クラスタのコア」か「クラスタの到達可能点」のどれかに分類し、各点のクラスタを決定していきます。
設定するパラメータはクラスタのコアと判定する最低隣接点数:minPts、隣接点と判定する距離閾値: epsです。
これらを使い 「コア点」: 距離閾値以内の点の数が最低隣接点数を満たす点 、「到達可能点」: コア点から距離閾値以内にあるが、最低隣接点数が満たない点 、「ノイズ」: 距離閾値以内にコア点が存在しない点 を決めます。
アルゴリズム
1.epsとminPtsの値を決める
2.ある点Xについて、他のすべての点からの距離を計算する
3.距離eps以内に存在する点はXに隣接する点とする
4.この点がminPts以上ある場合にクラスタとする
5.2-4を繰り返す
Pythonコード
ライブラリのインポートをします。DBSCANはsklearnに実装されています。
from sklearn.cluster import DBSCAN次に作成したデータXに対してDBSCANでクラスタリングを実施します。
DBSCANでは距離:eps、最小のサンプル数:min_samplesを設定しました。クラスタ数はクラスタリング結果から取得できます。
clustering = DBSCAN(eps=0.1, min_samples=5).fit(X)
result = clustering.labels_
n_clusters = len(set(result))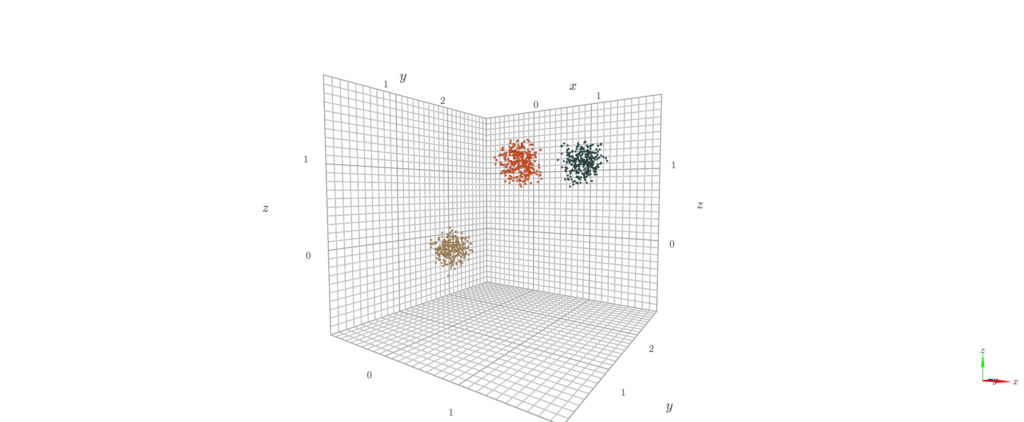
HDBSCAN
HDBSCANはDBSCANを階層型クラスタリングのアルゴリズムに変形したものになります。
DBSCANと比較して、異なる密度でもクラスタリングが可能であり、あらかじめ設定するハイパーパラメータはminPtsの一つのみになっています。
アルゴリズム
こちらの記事に詳しくてわかりやすい説明が載っています。
https://pberba.github.io/stats/2020/01/17/hdbscan/
大まかな処理は以下の通りです。
1.点ごとのコア距離を計算する
2.密度関数から2つの点がどのレベル(lamda)でつながるかをまとめた新たな指標Sを計算する
3.樹形図を作成する。これはlamda空間における単一連結クラスタリングと同等である。また、可能なすべてのレベルセットを繰り返し、クラスタを追跡することとも同じである
4.ハイパーパラメータminPtsによって樹形図を圧縮して複雑な樹形図を簡略化する
5.圧縮された樹形図木を用いて評価指標Sを最適化するように計算を行い、クラスタを抽出する
Pythonコード
HDBSCANのライブラリをインストールします。
pip install hdbscanインポートします。
import hdbscan次に作成したデータXに対してHDBSCANでクラスタリングを実施します。
HDBSCANでは最小のサンプル数:min_samplesを設定しました。クラスタ数はクラスタリング結果から取得できます。
clustering = hdbscan.HDBSCAN(min_samples=5).fit(X)
result = clustering.labels_
n_clusters = len(set(result))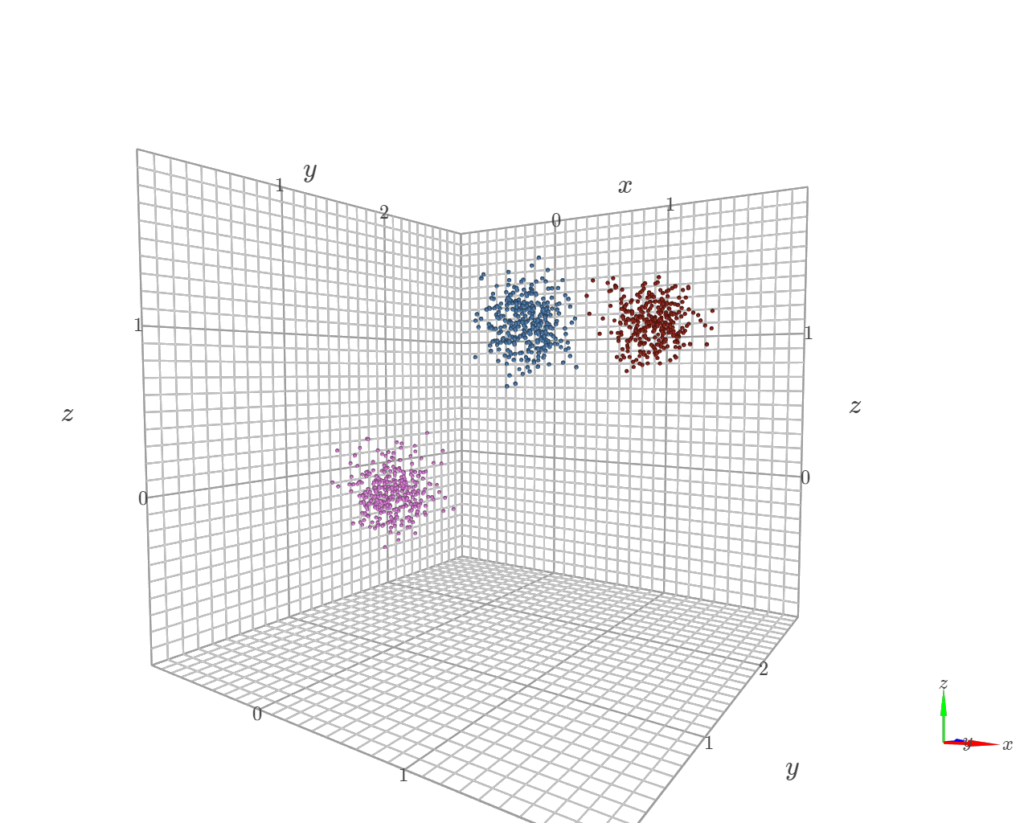
まとめ
3D点群に対してk-means, DBSCAN, HDBSCANでクラスタリングをしました。
k-meansはクラスタ数、DBSCANはepsとminPtsをあらかじめ設定しなければいけないのに対して、HDBSCANはminPts一つだけなので容易にクラスタリングできました。
今回のような小規模な3D点群データでは気になりませんが、どれも点の距離を計算するので大量の3D点群データに対しては処理時間がある程度かかってしまいます。








ディスカッション
コメント一覧
まだ、コメントがありません